IDENTIFICACIÓN GENÉTICA Y CONTROL DE FILIACIÓN EN RAPACES MEDIANTE TÉCNICAS MOLECULARES
|
Descripción |
La identificación genética y el control de filiación mediante técnicas moleculares en rapaces se basan en el análisis de marcadores de ADN de tipo microsatélite o STR y de tipo AFLP (Amplified Fragment Length Polymorphism). A partir de unas gotas de sangre o plumas se obtiene la huella genética, que es característica del animal y en la práctica irrepetible en la población a la que pertenece. Comparada con la de sus padres/hijos se puede contrastar la ascendencia/descendencia y llevar a cabo los denominados controles de filiación.
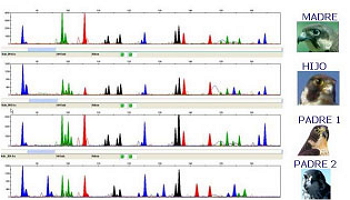
Contraste del perfil genético de un hijo frente al de dos posibles padres.
|
¿Cómo funciona? |
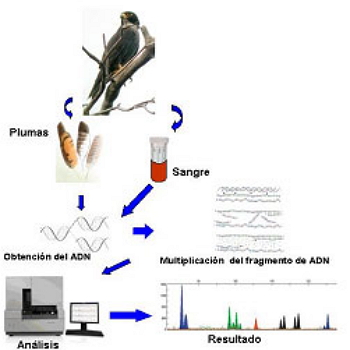
Representación esquemática de la metodología utilizada para la identificación genética.
El análisis de microsatélites es una técnica sencilla, rápida y de bajo coste que se puede realizar a partir de cualquier muestra que contenga células nucleadas (http://www.ucm.es/genetvet/identificacion-genetica-y-control-de-filiacion). Se procede al aislamiento del ADN y la posterior amplificación enzimática mediante la técnica de PCR. El análisis de estos fragmentos amplificados da lugar a lo que se denomina huella de ADN o huella genética, que es similar a un código de barras y es característica de cada animal.
Debido al polimorfismo de los marcadores, existe un gran número de combinaciones posibles de alelos, lo que hace muy difícil que dos individuos tengan la misma huella genética y, por lo tanto, es posible su diferenciación. Se trata de un método de identificación 'universal' muy potente, válido durante toda la vida del animal e infalsificable, por lo que en caso de pérdida, disputa o robo, haría posible la identificación de forma inequívoca.
El análisis genético de paternidad, pedigrí genético o control de filiación en rapaces, como en otras especies de animales, está basado en el contraste de la huella genética de un animal con la de sus ascendentes o progenitores. Para cada marcador, de los 2 alelos que presenta un individuo, uno lo ha heredado de la madre, (cuya veracidad en muchos casos no se cuestiona) y el otro del padre biológico o verdadero. En el caso de los AFLPs, cada banda existente en la huella genética de un individuo, debe estar presente en uno de sus dos progenitores. Si se realiza este análisis para un conjunto de 8-10 marcadores, la potencia de exclusión de paternidad es muy elevada, próxima al 100%. Si el presunto padre es rechazado como padre biológico el resultado es categórico y absoluto.
|
Ventajas |
La determinación de la huella genética o huella de ADN permite tanto identificar a los animales mediante un método universal altamente polimórfico e infalsificable, que perdura toda la vida del animal, como verificar o certificar la ascendencia o descendencia de los animales por comparación con la huella genética de sus parentales o descendencia.
La escasa muestra biológica necesaria (basta con una gota de sangre), el elevado polimorfismo de estos marcadores frente a otros utilizados anteriormente, y la facilidad de determinación de forma inequívoca, hace que sean técnicas muy utilizadas de forma rutinaria para el registro de animales en Libros Genealógicos de las distintas razas, para verificar su identidad y garantizar la pureza racial.
La facilidad, rapidez de determinación y bajo coste, la hacen idónea para la generalización en Comunidades Autónomas que realizan el seguimiento de los ejemplares de especies autóctonas criadas en cautividad, criadores, y particulares de distintos animales y los técnicos del SOIVRE que velan por la aplicación del Real Decreto 1739/1997, sobre medidas de aplicación del Convenio sobre comercio internacional de especies amenazadas de fauna y flora silvestres (CITES), hecho en Washington el 3 de marzo de 1973 y del Reglamento (CE) nº 338/97 del Consejo, de 9 de diciembre de 1996, relativo a la protección de las especies de la fauna y flora silvestres mediante el control de su comercio.
|
¿Dónde se ha desarrollado? |

Servicio de genética.
Esta tecnología ha sido desarrollada por el Servicio de Genética de la Facultad de Veterinaria de la Universidad Complutense de Madrid por un grupo investigador con una dilatada participación en proyectos de investigación nacionales y europeos que les han permitido desarrollar las metodologías necesarias para esta oferta.
Este centro viene ofreciendo sus servicios desde el año 1996. Desde su puesta en marcha, la demanda de la identificación genética y los controles de filiación ha surgido tanto de particulares como de instituciones, como la Consejería de Medio Ambiente de la Comunidad de Madrid, de la Junta de Castilla y León, de CITES o del SEPRONA, lo que indica el amplio rango de posibilidades que ofrece.
La base tecnológica de este Servicio de Genética está apoyada en las investigaciones que sus autores han llevado a cabo durante los últimos 20 años, y difundido a través de numerosas publicaciones científicas en revistas relevantes, contando, además, con gran experiencia en colaboraciones con empresas y asociaciones. Forman parte del Grupo de “Nutrigenómica Animal” del Departamento de Producción Animal de la Facultad de Veterinaria de la UCM, dirigido por Susana Dunner. Este grupo de investigación está incluido en el clúster de Agroalimentación y Salud del Campus de Excelencia Internacional CEI-Moncloa.
[más información sobre el departamento y el grupo de investigación]
|
Y además |
El Servicio de Genética de la Facultad de Veterinaria de Madrid, ofrece servicios de apoyo a las actividades clínicas diarias de los veterinarios (controles de paternidad, identificación genética, sexado de aves mediante técnicas moleculares, diagnóstico de portadores de enfermedades hereditarias...), y a otros profesionales con otros fines (valoraciones genéticas, estimación de parámetros genéticos, etc.).
|
Contacto |
|
© Oficina de Transferencia de Resultados de Investigación – UCM |
|
Descargas PDF |
|
Clasificación |
|
Investigador responsable |
Javier Cañón Ferreras: genetica@vet.ucm.es
Departamento: Producción Animal
Facultad: Veterinaria